Bier, statistisch gesehen
Die nach dem bayerischen Reinheitsgebot einfache Zusammensetzung von Bier täuscht darüber hinweg, das sich komplexe Zutaten während des Brauvorgangs molekular verändern.
Das bayerische Reinheitsgebot ist mittlerweile über 500 Jahre alt und schreibt vor, dass Bier nur aus den Zutaten Hopfen, Malz, und Wasser gebraut werden darf. Diese recht einfach erscheinende Zusammensetzung von Bier täuscht aber über die Tatsache hinweg, dass es sich dabei ‚molekular‘ um sehr komplexe Zutaten handelt und diese während des Brauvorgangs und der anschließenden alkoholischen Gärung noch vielfältigen weiteren (Fermentations-) Prozessen unterzogen werden.
Non-Target Screening (NTS)
So sind neben hochmolekularen Proteinen (z. B. in Ihrer Funktion als Enzyme) und langkettigen Zuckermolekülen, vor allem auch eine Vielzahl kleiner Metabolite und sekundärer Pflanzeninhaltstoffe enthalten, die – in Farbe, Geschmack und weiteren Eigenschaften resultierend – in großer Varianz im Bierbrauprozess erhalten bleiben oder aber erst entstehen.
Konsequenterweise lassen sich im Bier auch Hunderte von bekannten Substanzen detektieren [1]. Daneben befinden sich aber auch noch unzählige weitere, meist unerwartete oder unbekannte Verbindungen im Bier. Für eine umfassendere Analyse und Bewertung von solch komplexen Proben, wie z. B. Bier, kann man heute neue Wege beschreiten und sich des massenspektrometrischen Non-Target Screenings bedienen.
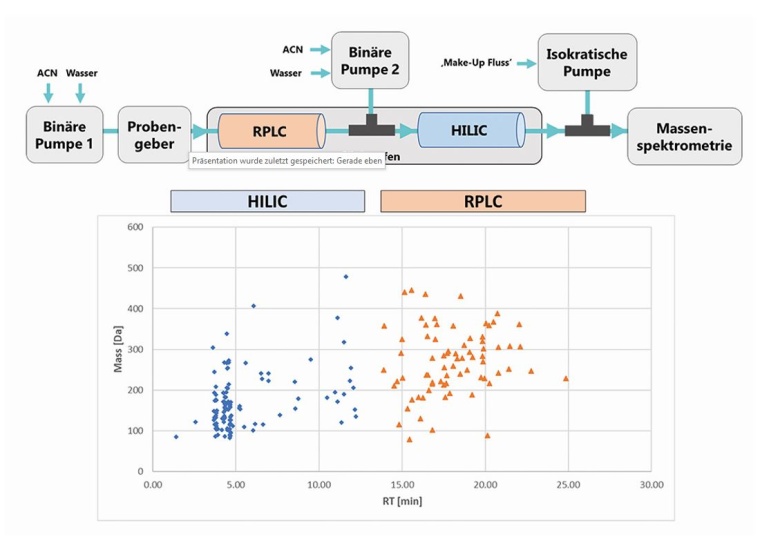
Non-Target Screening (NTS) für das Umkehrphasenflüssigchromatographie in serieller Kopplung mit hydrophiler Interaktionsflüssigchromatographie und hochauflösender und akkurater Massenspektrometrie (RPLC-HILIC-HRMS) kombiniert wird, stellt seit nunmehr über zehn Jahren eine bestehende Lösung dar, um komplexe Proben zu analysieren [2]. Diese eröffnet neue Blickwinkel und eignet sich unter anderem zur Qualitätssicherung von Wasser [3] und Authentizitätsbestimmung von Getränken und anderen Lebensmitteln auf molekularer Ebene [4].
In diesem Artikel geht es aber weder darum, enthaltenen (unbekannten) Bier-Komponenten einen Namen zu geben, noch darum Verunreinigungen bzw. Pestizide zu suchen, noch anzugeben, welche gefunden worden sind. Im Gegenteil, es werden im Folgenden Darstellungen präsentiert, die aus der statistischen Handhabung von analytisch erfassten Daten resultieren. Diese Perspektive eröffnet eine vollständig andere Sichtweise auf komplexe Proben und deren grundlegenden Eigenschaften.
Molekulare Fingerabdrücke
Im Gegensatz zur heute herkömmlichen quantitativen Analytik, in der man bekannte (und meist als Referenzsubstanz vorliegende) organische Moleküle untersucht und deren Menge bestimmt, basiert die Datenauswertung im Bereich der NTS zunächst auf der Erfassung analytischer Daten und deren Transfer in korrespondierende physikalisch-chemische Parameter. Solche Parameter stellen die roten und blauen Signale in Abbildung 1 dar. Analysiert man unterschiedliche Bierproben mit der oben erwähnten RPLC-HILIC-HRMS, erhält man solche oder ähnliche „Fingerabdrücke“ von Proben, bestehend aus einer Vielzahl von Merkmalen, sogenannten Features. Diese Features, bestehen aus der erfassten chromatographischen Retentionszeit, der detektierten Masse, eventuelle Massenfragmente und der jeweiligen Signalintensität(en). Sie spiegeln eindeutige Komponenten bzw. organische Moleküle wider und können anschließend oftmals über den Abgleich mit analytischen oder stoffbezogenen Datenbanken identifiziert und bei Möglichkeit durch den Vergleich mit Referenzmaterialien validiert werden.
Für die eindeutige Charakterisierung von Proben ist eine Identifizierung der Moleküle aber nicht immer zwingend notwendig. Schon allein durch den Vergleich von detektierten Features in verschiedenen Proben bzw. die Unterschiede in deren Signalintensitäten können Proben charakterisiert, unterschieden und letztlich bewertet werden. So zeigen die Abbildungen 2,3 und 4 klassische Auftragungen von Volcano-Plots (Abb. 2), der PCA (Abb. 3) und Box-Plots der Features von unterschiedlichen exemplarischen alkoholfreien Weißbieren (W1-W6) und Pilsnern (P1 und P2).
Im Volcano-Plot sind die Signalintensitätsunterschiede verschiedener Features zwischen zwei Proben, sowie deren P-Wert (t-Test) aufgetragen. Je weiter oben und je näher an einer der beiden Seiten ein Feature liegt, desto charakteristischer ist dieses für die jeweilige Probe. In Abbildung 2 sind die Features von einem Pilsner und eines Weißbiers gegeneinander aufgetragen. Die Features auf der linken Seite der Abbildung (farblich hervorgehoben) sind hier charakteristisch für das Pilsener, die auf der Rechten für das Weißbier. Bei Erweiterung des Probenkollektives kann man auf diese Weise Features d.h. Moleküle erfassen, die generell signifikant in Pilsnern oder Weißbieren auftreten. Bei der späteren Analyse einer zunächst unbekannten Probe könnte man darauf aufbauen und einordnen, ob es sich um ein Pilsner oder ein Weißbier handelt.
Marker-Molekül-Analyse
Dass sich Pilsner und Weißbiere grundsätzlich analytisch unterscheiden lassen, zeigt sich auch mittels Hauptkomponentenanalyse (PCA). In Abbildung 3 ist eine solche PCA über sechs Weißbiere und zwei Pilsner Biere (jedes Bier wurde hier aus statistischen Gründen dreimal analysiert). Die Pilsner unterscheiden sich in Hauptkomponente 2 von den Weißbieren und lassen sich somit deutlich von den anderen Bieren unterscheiden. Solche Unterschiede können auch auf der Ebene einzelner Features untersucht werden. Abbildung 4 zeigt die Intensitäten eines beispielhaften Features über alle untersuchten Bierproben hinweg. Das hier gezeigte Feature liefert in den Pilsnern signifikant höhere Intensitäten als in den Weißbieren. Somit könnte dies vielleicht als ein sogenanntes Marker-Molekül für Pilsner dienen. Selten sind aber einzelne Marker-Moleküle allein zielführend für die eindeutige Aussage über Qualität und Zuordnung. In der Kombination vieler solcher signifikanter Marker-Moleküle allerdings nimmt die statistische Sicherheit zu und die Belastbarkeit der Aussage verhärtet sich. Wenn dann solche Marker-Moleküle in weiteren Analysen und in größeren Probenkollektiven statistisch abgesichert werden können, wird im nächsten Schritt versucht, die Marker-Substanzen, wie oben beschrieben zu identifizieren. Dieses Vorgehen würde bei der Einordnung der Moleküle helfen, aber keinen Unterschied bei der Relevanz der Features machen. Auf der anderen Seite wird die statistische Belastbarkeit umso besser, je mehr Proben vermessen werden. Aus diesem Grund und zum Zweck der Verbesserung der Datenlage werden derzeit eine Vielzahl von Bieren aus Brauereien und von Konsumenten bezogen. Die daraus gewonnenen Features werden zusammen mit weiteren Informationen zu den Bieren in Datenbanken hinterlegt. So wird der molekulare Fingerabdruck gesichert und zur retrospektiven Datenanalyse vorgehalten und dient den Brauereien auch als „molekulare Grundlage“ und als ihr „status quo“.
Zusammenfassung
Es ist festzuhalten, dass das massenspektrometrische Non-Target Screening über die analytische Ermittlung von probenenthaltenen Molekülen (wie in diesem Falle in alkoholfreien Bieren) und deren Übersetzung in physikalisch-chemische Parameter sehr klare Vergleiche und statistische Interpretationen zulassen. Mit diesen sogenannten Features lassen sich somit eindeutige Authentizitätsbestimmungen sowie Qualitätskontrollen durchführen. Dafür ist es auch nicht nötig, die relevanten Substanzen alle beim ‚Namen zu kennen‘, d. h. dass diese nicht identifiziert werden müssen. Lediglich die statistische Grundlage muss eindeutig sein und die Marker-Moleküle zuordenbar. Die Statistik ist in diesem Fall der Schlüssel zu neuen Erkenntnissen und zukunftsweisenden Sichtweisen.
Es ist offensichtlich, dass dieser Ansatz nicht nur im Bier funktioniert, sondern auch mit anderen Getränken sowie in weiteren Lebensmitteln und Produkten mit eindeutigem molekularem Fingerabdruck.
Danksagungen
Ein sehr großes Dankeschön geht an Martin Zarnkow und Andreas Laus vom Forschungszentrum Weihenstephan für Brau- und Lebensmittelqualität (der Technischen Universität München) für die Überlassung der großen Zahl alkoholfreier Biere für diese Publikation und der fachlichen Diskussion rund um das Thema Bier.
Ein ebenso großes Dankeschön geht an unsere Kooperationspartner in instrumenteller NTS-Analytik aus dem Hause Thermo Fisher Scientific. Deren tatkräftige Unterstützung zur Weiterentwicklung und den Übertrag der NTS-Analytik in die Routine unter Aufrechterhaltung der Flexibilität und der langfristigen Datennutzung ist vorbildlich und nachhaltig.
Autoren: Stefan Bieber und Thomas Letzel, Analytisches Forschungsinstitut für Non-Target Screening